이번 파트에서는 Opensim 환경과 Task 에 대해 설명하도록 하겠습니다. (competition이 종료된 후 새로운 webinar 가 나왔습니다. 자세히 설명되어 있으니 참고 바랍니다.)
I. Opensim
강화 학습에서 환경(environment)이란 매우 중요한 요소입니다. 강화학습 Agent가 interaction을 통해 주어진 task를 수행하는 공간이 바로 이곳이기 때문에 당연한 말이겠죠. 어떤 방식으로 접근하던 환경에 대한 깊은 이해가 선행되어야만 좋은 솔루션이 나올 수 있다해도 과언이 아닙니다. 때문에 저희 팀은 이번 컴페티션을 진행하면서 opensim이라는 환경을 심도 있게 이해하기 위해 많은 노력을 기울였습니다.
Opensim은 신경근 골격 모델(neuromusculoskeletal model)의 움직임을 시뮬레이션 하기위한 소프트웨어입니다. 실험데이터를 분석하고 움직임을 시뮬레이션하여 해부학, 생리학, 운동역학 등의 접근을 가능하게 합니다. 굉장히 다양한 분야에서 쓰일 수 있는데, 운동 시퀀스를 분석해서 개선한다든가, Modeling Evaluation and Control Optimization of Exosuit with OpenSim 와 같이 의족이나 기타 보조기기 착용 시 움직임 예측 등등에 쓰입니다. 시스템의 모든 부분을 전부 언급하기에는 너무나 방대하므로 여기서는 개괄적인 부분과 opensim-rl에 관련된 부분 위주로 기술하도록 하겠습니다.
Workflow
우선 opensim을 이용한 기본적인 분석 시퀀스는 다음과 같습니다.
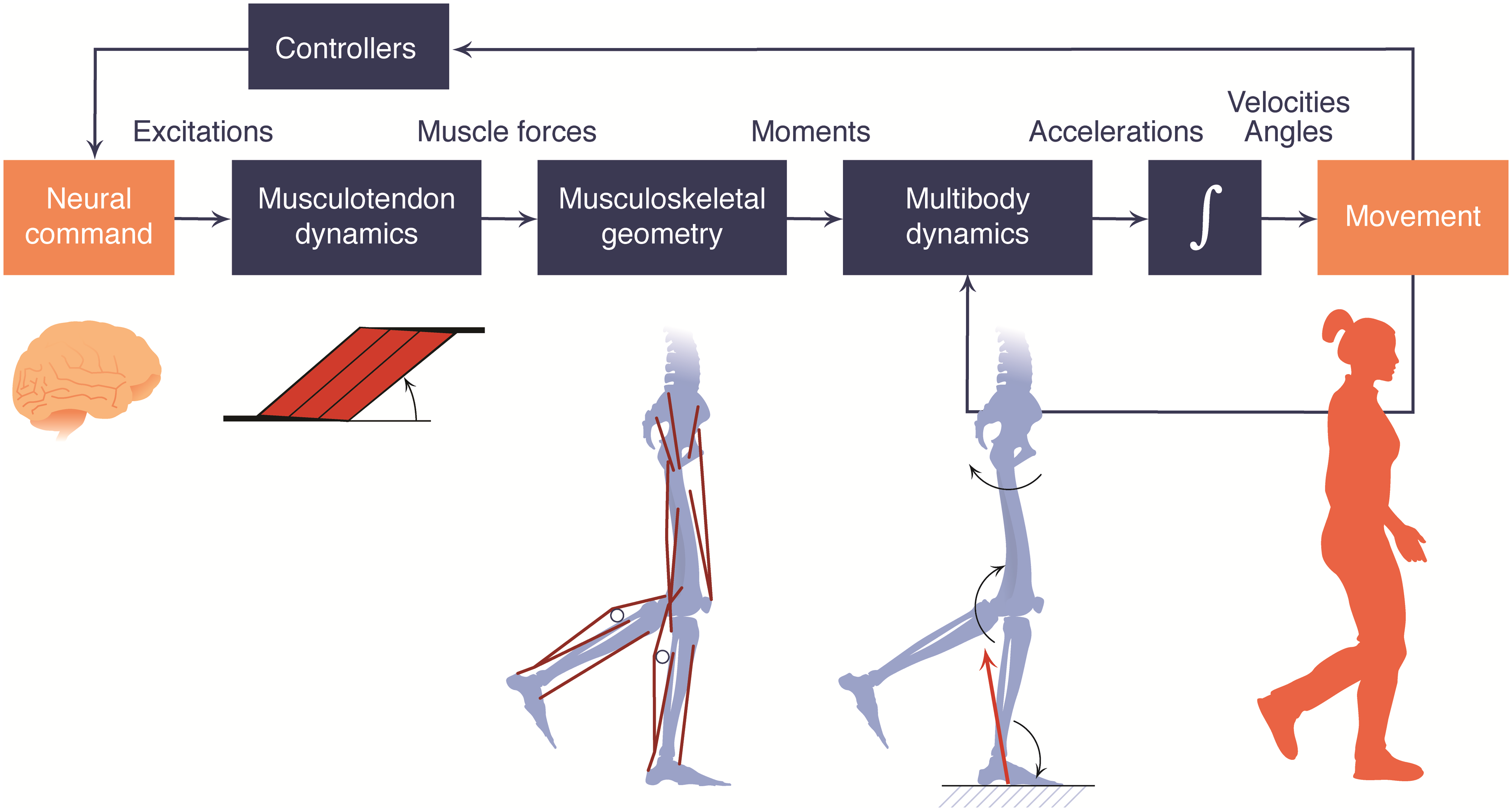
from OpenSim: Simulating musculoskeletal dynamics and neuromuscular control to study human and animal movement
방대한 시스템답게 생체역학적 움직임에 대한 다양한 분석이 가능한데요. 어떤 방향으로 문제를 보는가에 따라 크게 2가지로 나뉠 수 있습니다.
- Inverse problem
- Forward problem
1. Inverse problem
일반적으로 opensim을 쓰는 많은 생체 역학 분야의 연구자들이 쓰는 방식입니다. 실험데이터를 통해 움직임에 관련된 여러 가지 분석을 하기 위해 사용되죠. 위의 시퀀스 그림에서 보면 Movement -> Neural command 방향으로 진행되는 프로세스라고 볼 수 있습니다. 마커를 부착해서 실험으로부터 특정 모션에 관한 데이터를 얻어내고, 이를 통해 움직임의 역학 모델을 만들어 근육의 활성화 정도와 근육을 활성화 시키는 데 필요한 신경의 흥분 정도를 계산합니다. 다음과 같은 문제들의 답을 얻어내는 과정을 통해 motion에 관련된 여러 가지 수치를 얻을 수 있습니다.
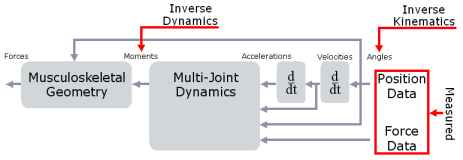
Inverse kinematic
해당 motion을 만들어내는 각 joint의 angle 값들을 구합니다. pose와 관계된 내용이므로, 질량이나 관성 같은 값들은 필요 없습니다.
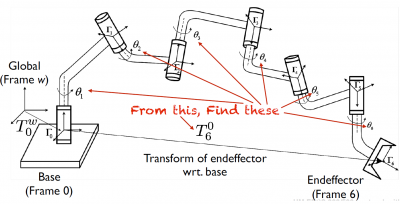
CF) Forward kinematics VS Inverse kinematics
- Forward kinematics는 주어진 joint position(angles)들로부터 말단장치(end effector)의 위치를 구합니다.
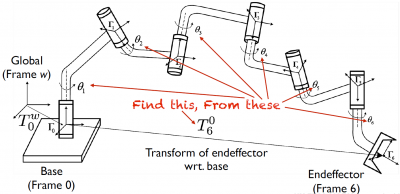
- Inverse kinematics는 주어진 말단장치(end effector)의 위치로부터 joint position(angles)들을 구합니다.
Inverse dynamics
해당 motion을 유발하는 force와 moment값들을 구합니다. 여기서 얻어낸 force들을 사용해서 어떤 근육들이 얼마만큼의 힘을 받는지 알아냅니다. 이를 계산하기 위해 질량과 관성 같은 값들이 필요합니다.
기본적으로 뉴턴과 오일러의 수식이 사용됩니다.
- Newton (linear): F = m.a (Force = mass x linear acceleration)
- Euler (angular): M = I.α (Moment = mass moment of inertia x angular acceleration)
그리고 다음 가정들이 적용됩니다.
- Joint는 무마찰 핀 조인트(frictionless pin joint)
- 질량 중심에 집중질량이 있다고 가정된 각 강체들의 조합
- 주동근과 길항근( agonist and antagonist)의 공동(동시)수축은 없음
- 공기 저항은 최소
완전히 정밀한 인체 동작 연구까지는 불가능하지만, 상당히 유의미한 결과를 얻을 수 있습니다.
2. Forward problem
이번 competition에서 쓰이는 opensim-rl에서 사용되는 방식입니다. Neural command -> Movement 방향으로 진행됩니다. 일반적인 인간, 동물의 움직임을 떠올리면 됩니다. neural command의 excitation 정도에 따라 근육의 수축/이완의 길이와 속도가 결정되고 그에 따른 motion이 만들어집니다.
Architecture
Opensim의 전체 구조는 다음 그림과 같습니다. 하부 단은 simtk API 를 이용하여 이루어져 있는데 실제 시뮬레이션 관련 계산은 대부분 여기서 이루어집니다.
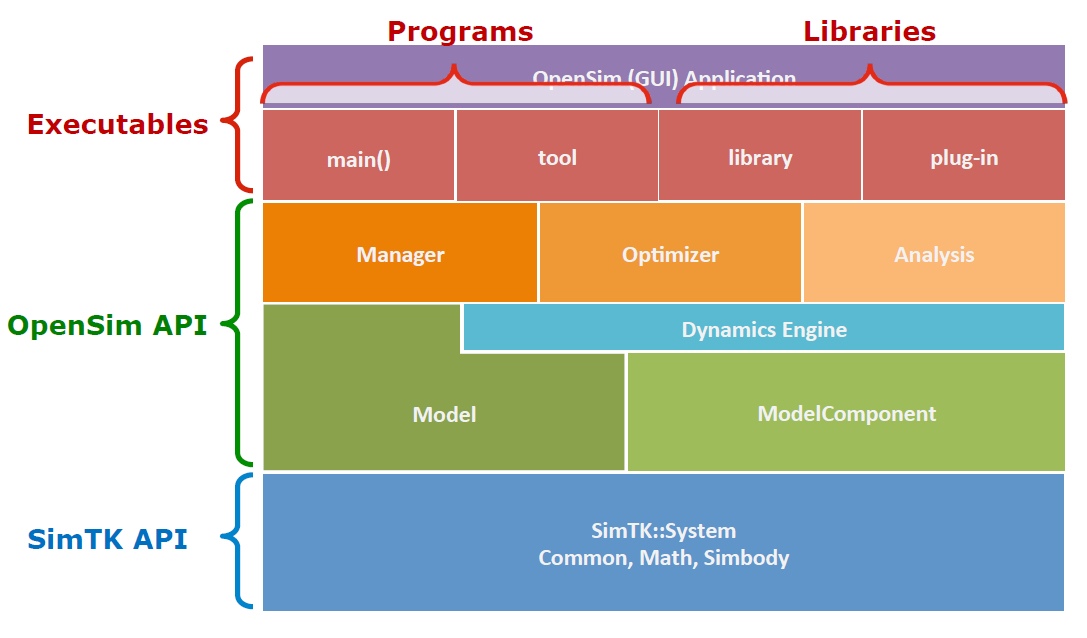
Exacutables
Opensim API를 이용한 여러 가지 응용 시스템들입니다. opensim gui app부터 이번 competition에서 사용되는 opensim-rl 까지 모두 이 부분에 속합니다.
대략 다음과 같은 인터페이스를 통해 opensim을 사용 가능합니다.
- GUI
- command line
- XML
- Python scripting
- MATLAB scripting
- C++
Opensim API
시뮬레이션 수행을 위한 모델의 구조를 정의합니다. 근골격 관련 ,신경 관련 등의 설정 관련 모듈들을 갖고 있습니다. SimTK를 이용하여 시뮬레이션을 수행합니다.
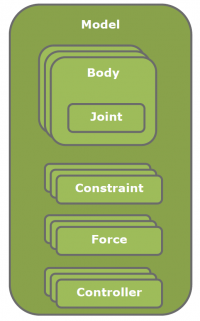
Model: 근골격 모델 class. xml 파일을 통해 설정을 읽어올 수 있습니다. ModelComponents 들을 포함합니다.
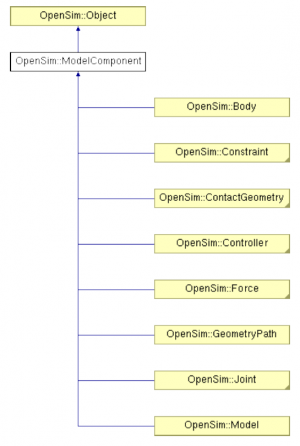
OpenSim Model Class Structure. from OpenSim’s Architecture and API ModelComponent: 근골격 모델의 특정 부분을 나타내는 class. simtk의 multibodySystem에서 사용되어 계산됩니다.
OpenSim ModelComponent. from OpenSim’s Architecture and API Manager: 전체 시뮬레이션 동작을 관리하는 class. SimTK::Integrator 와 SimTK::TimeStepper를 이용하여 시뮬레이션을 수행합니다.
Analysis: 분석 관련 plug-in class. 시뮬레이션과 관련된 여러 가지 분석된 수치들을 기록할 때 사용합니다.
SimTK
시뮬레이션 동작을 위한 연산 엔진입니다. SimTK::Integrator 와 SimTK::TimeStepper이 time step 별로 시뮬레이션의 변화를 계산합니다. 내부적으로 봤을 때 그림과 같이 subsystem 들로 구성되어 있습니다.
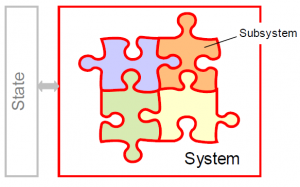
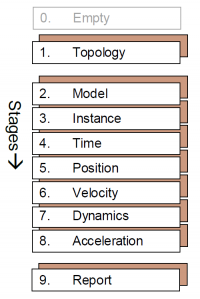
- t: time
- q: generalized coordinates
- u: generalized speeds
- z: auxiliary variables
Structure
자 이제 위에서 언급했던 모든 모듈들과 시스템들이 합쳐진 전체 구조를 봅시다. opensim에서 visualization되는 model부터 하부의 simtk까지 다음 그림과 같은 연결 구조로 되어 있습니다.
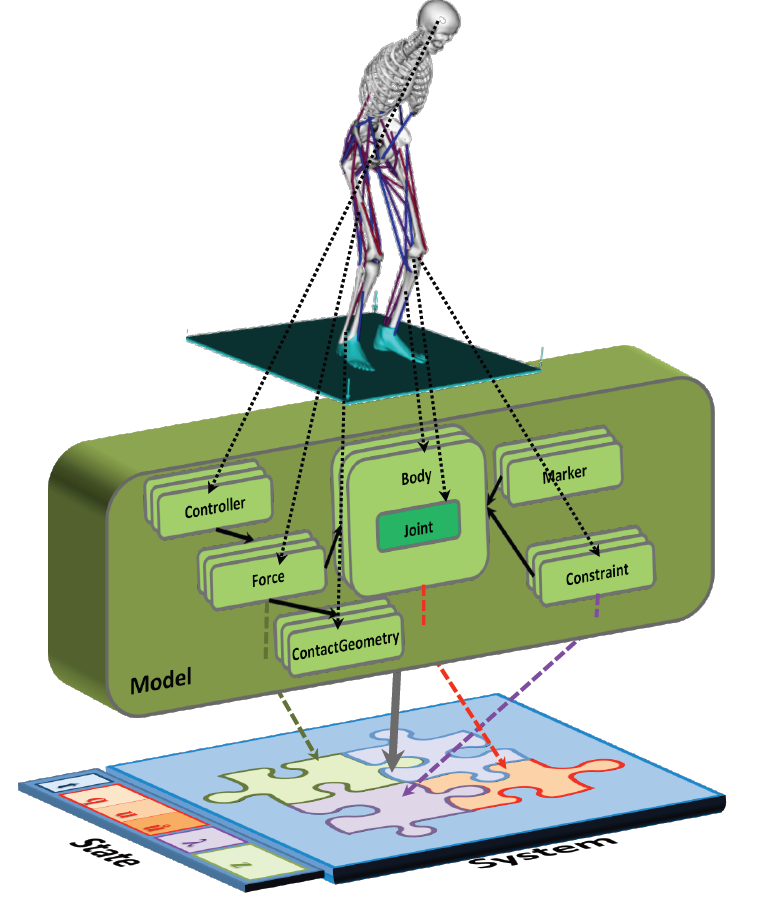
Tools
Opensim은 필요한 수치들을 구할 수 있도록 미리 구현된 application인 computational tool들을 제공해줍니다. Opensim GUI app에서 사용할 수 있고, script 방식으로 matlab이나 python에서도 직접 함수를 호출하여 사용 가능합니다.
tool은 기본적으로 필요한 설정파일, 데이터 파일들을 load해서 사용하는데요. RRA tool의 예제를 보면 다음과 같습니다.
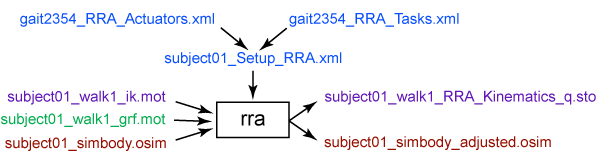
GUI app에서의 사용법은OpenSim: Complete Run-through youtube를 참고하시기 바랍니다.
또한 라이브러리 형태로 제공된 tool들을 코드상에서 사용하는 예제는 medipixel git 을 참고하시기 바랍니다.
Scale
실험 데이터는 보통 모션캡쳐를 통해 얻게 됩니다. 신체 여러 부위의 마커를 달고 측정되는 각각의(x, y, z)좌표를 기록하는데요, 여기서 획득하게 되는 좌푯값들은 마커를 달고 있는 사람의 신체비율에 따라 조금씩 달라집니다. 따라서 정확한 시뮬레이션을 위해서는 실제 측정한 사람의 신체 비율과 시뮬레이션에 사용될 모델의 비율을 맞춰주는 작업이 필요한데, 이 단계에서 사용되는 tool이 Scale tool입니다.
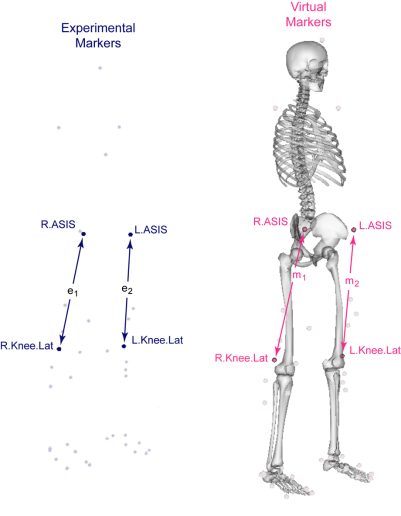
Inverse Kinematics
IK(Inverse Kinematics) tool은 각 실험 데이터로부터 opensim model의 포즈를 만들기 위해 사용됩니다. 각 time step 별로 측정된 marker의 좌푯값과 가장 일치하는 포즈로 모델을 위치시키는 일반화된 좌표(generalized coordinate)값들을 계산합니다. 일치하는 포즈는 모델의 마커와 실험데이터 마커의 거리인 marker error를 최소화하는 방향으로 계산하는데요. marker의 중요도에 따라서 가중치를 부여합니다. 그래서 각 marker들의 weighted least square값을 합하여 이것의 min 값이 되는 포즈를 찾아냅니다. 수식으로 표현하면 다음과 같습니다.
$$ min_q \left[ \sum_{i\in{markers}}w_i |x_i^{exp} - x_i(q)|^2 \right] $$
Residual Reduction Algorithm
실험 데이터는 noise를 포함합니다. noise 생기는 이유는 여러 가지가 있으나 가장 중요한 요인은 크게 3가지가 있습니다.
- 팔이 없다
- 측정상의 noise
- 측정 위치 마커 에러
이러한 요인들에의해 inverse dynamics를 수행할때 residual force라는 추가적인 힘이 발생하게 됩니다. 그렇기 때문에 전체 inverse dynamics를 통해 구하여진 force에서 residual force를 근접(최소화하여)하게 산출 해서 빼줘야 정확한 force를 얻어 낼 수 있습니다. 아래식과 같이 나타내게 됩니다.
$$ F + F_{\text{residual}} = ma $$
RRA tool은 이런 기능을 수행합니다.
Static Optimization
Inverse dynamics를 dicrete time별로 풀어냅니다. 여기서 알아낸 force와 moments를 사용하여, 근육의 활성화 정도를 계산합니다. 여기서 활성화 정도를 계산할 때 근력이 가지고 있는 한계를 넘어가지 않도록 제한합니다. 수식을 보면 다음과 같습니다.
$$ \sum_{m=1}^n(a_mF_m^0)r_{(m,j)} = \tau_j $$
- $a_m$: 근육의 activation level
- $F_m^0$: 최대 수축 힘
- $r_{(m,j)}$: moment arm
이를 force-length-velocity 속성을 추가하여 바꾸면 다음과 같습니다.
$$ \sum_{m=1}^n[a_mf(F_m^0,l_m,v_m)]r_{(m,j)} = \tau_j $$
- $f(F_m^0,l_m,v_m)$: force-length-velocity이용한 힘 관련 함수
- $l_m$: 근육의 길이
- $v_m$: 수축 속도
Computed Muscle Control
아래와 같이 Static Optimization과 Forward dynamics를 번갈아 수행하며, 해당 kinematics를 만들어낼 수 있는 근육의 activation level을 계산합니다.
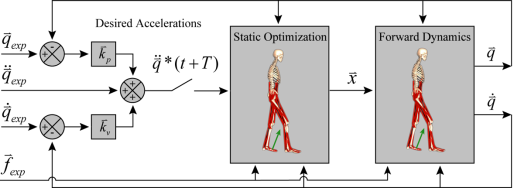
Forward dynamics
Forward problem을 수행합니다. 일반적으로 전통적인 opensim 실험데이터에는 신경의 excitation은 포함되어 있지 않습니다. 따라서 아래와 같은 tool들을 차례대로 이용하여 얻어낸 excitation(control) 값을 사용합니다.
- Scale
- Inverse Kinematics
- Residual Reduction Algorithm
- Computed Muscle Control
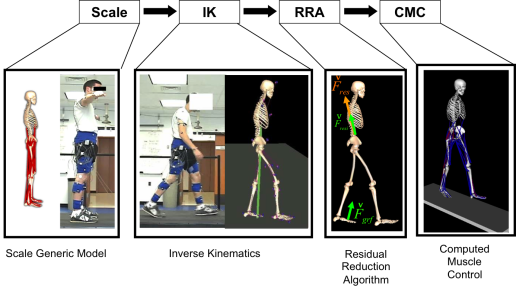
II. Opensim-rl
Opensim-rl은 아래 그림에서 main()과 같이 opensim 모델과 manager를 이용하여 시뮬레이션을 수행하는 프로그램입니다.
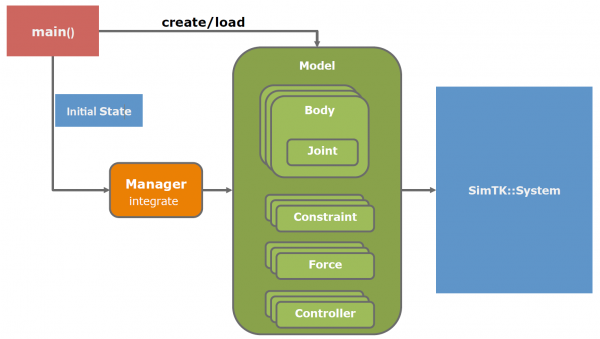
swig를 사용하여 python scripting 방식으로 구현되어 있습니다. 위의 설명에서 forward problem 과 같은 방향으로 진행됩니다. agent는 neural command(action)을 이용하여 model을 컨트롤 합니다.
III. Walking/Running gait
이번 competition에서 주어진 Task는 다음과 같습니다.
- Round1: 3초간 3m/s에 맞춰 뛰기
- Round2: 10초간 1.25m/s에 맞춰 걷기 1
저희 팀은 이 Task 해결을 위해 환경파악 못지않게 “Walking/Running gait” 문제에 관한 탐구에도 많은 시간을 투자하였습니다.
강화학습에서 Task가 파악이 잘되었다면 reward 설정도 잘할 수 있습니다. Task를 잘 파악한다면 agent 학습 시 그 목적에 따라 어떤 부분을 reward로 설정하여 더 격려할지 아니면 penalty로 설정하여 억제할지 명확해지기 때문에, 원활한 학습이 가능해집니다. 또한, 다음 part에서 언급할 imitation learning과 같은 방법론을 사용할 때에도 Task에 대한 명확한 이해가 있어야만 가이드로 사용할 demonstration이 잘되었는지 안되었는지에 대해 알 수 있습니다. 운전을 잘 알려주려면 운전에 대해 잘 알아야 하고, 달리기를 잘 알려주려면 달리기를 잘 알아야 합니다. Task에 대한 이해도가 높으면 높을수록 좋은 agent를 만들 수 있는 확률이 올라갑니다. 그런 의미에서 걷기와 달리기 메커니즘에 대한 이해는 매우 중요한 부분이었습니다.
Experimental Data
마침 nips2018에서는 작년과는 다르게 experimental data를 사용할 수 없다는 제약사항이 사라졌습니다. 우선 simtk 커뮤니티를 검색하면서 여러 공개데이터 셋과 기존 연구들을 조사하였는데요, 가장 중점적으로 확인한 사항은 gait cycle 중 kinematic의 변화들, 즉 joint들의 움직임이 어떤 패턴을 갖는가였습니다. 다행히 이것에 대해서는 여러 가지 그래프와 데이터의 형태로 많은 분석이 진행되어 있었습니다. 그 중 Multiple Speed Walking Simulations라는 연구에서 round 2에서 사용할만한 데이터셋을 발견할 수 있었습니다. walkning speed가 정확하게 1.25m/s는 아니었지만 subject 2, 3의 free speed의 경우에는 그와 비슷한 속도(1.21, 1.29)로 사용할만한 수치였습니다. round1은 opensim-rl page에 예시로 제공된 Muscle contributions to mass center accelerations over a range of running speeds 연구에서 제공된 데이터셋이 정확하게 3m/s로 달리는 데이터였기 때문에 비교적 쉽게 결론을 내릴 수 있었습니다.
위의 데이터셋을 opensim에서 동작시키면 다음과 같은 모습입니다. 먼저 round1 용 데이터는 다음과 같습니다.


Proper data-type
※ 아래 나오는 Imitation learning 관련 자세한 내용 설명은 Imitation learning posting에서 다룰 예정입니다.
그러고 나서 했던 고민은 이 데이터들을 어떻게 하면 imitation learning에서 사용할만한 demonstration형태로 바꿀 수 있는가였습니다. 최초의 시도는 완전한 Behavioral cloning을 생각 하였기 때문에, action에 관련된 데이터도 만들어내기 위해 노력하였습니다. 위 opensim tool에서 설명하였지만, 기존 opensim을 이용한 연구자들이 forward problem을 해결하는 방식처럼 Data -> CMC tool 과정을 거쳐 Neural command 즉 action을 만들어내려고 했지요. 다만 이 시도는 몇 가지 제약사항 때문에 난관에 봉착하였는데
- 기존에 연구된 데이터들은 각기 다른 opensim model들을 사용했다
- 사용된 model에 따라 관절의 자유도(DoF: Degree of Freedom)가 다르다
- 사용된 model에 따라 근육의 개수가 다르다
- CMC tool을 사용할 때 보통 external forces, reserve actuators를 등록하여 사용하는데, opensim-rl환경에서는 사용 불가능이다
결국, 이런 문제점들 때문에, Behavioral cloning으로 문제를 해결하려는 접근은 폐기되었습니다.
그래서 action에 대한 data 없이 관찰되는 모습(kinematics)의 data만 이용하여 학습할 수 있는 방법론2을 찾아내었고, 실험데이터를 적합한 demonstration으로 가공하기 위해 고민하였습니다.
Making Demonstration
실험데이터는 기본적으로 다음과 같은 제약들이 있었습니다.
- Round1은 3초간 동작, Round2는 10초간 동작해야 함에 비해 demo 시간이 너무 짧다(1초 이하)
- 제자리 혹은 짧은 거리만 이동한다
- 스타팅 동작이 없다
위 제약은 kinematics 데이터를 수정하여 해결하였습니다. 실험데이터에 있는 gait cycle(걷는 동작)을 반복적으로 이어붙이고, 중간에 어색한 부분은 데이터를 새로 만들어 넣었습니다.
그렇게 만들어진 round 1의 Demo는 다음과 같았습니다.


또한, 해결해야 했던 부분은 여기서 만들어진 kinematic 데이터의 형태와 opensim-rl에서 관측되는 observation state의 형태가 다르다는 점이었습니다.
이를 해결하기 위해 opesim python script를 이용하여 간단한 transfer 프로그램을 만들었습니다. 해당 코드는 medipixel git에서 확인 가능합니다. 변환과정을 간단하게 설명하면
- motion 데이터를 읽어온다
- 0.01 초마다 아래 과정을 반복
- motion 데이터의 time step 별로 저장된 데이터를 통해 시뮬레이션을 구동시킨다
- model의 정보(observation)를 읽어와서 list에 저장한다
- list에 저장된 정보의 shape을 변환한다
위와 같은 과정을 거쳐 새로운 demonstration으로 사용 가능한 형태로 변환시킵니다.
References
- Seth, A., Hicks, J. L., Uchida, T. K., Habib, A., Dembia, C. L., Dunne, J. J., … & Hamner, S. R. (2018). OpenSim: Simulating musculoskeletal dynamics and neuromuscular control to study human and animal movement. PLoS computational biology, 14(7), e1006223.
- Delp, S. L., Anderson, F. C., Arnold, A. S., Loan, P., Habib, A., John, C. T., … & Thelen, D. G. (2007). OpenSim: open-source software to create and analyze dynamic simulations of movement. IEEE transactions on biomedical engineering, 54(11), 1940-1950.
- Liu, M. Q., Anderson, F. C., Schwartz, M. H., & Delp, S. L. (2008). Muscle contributions to support and progression over a range of walking speeds. Journal of biomechanics, 41(15), 3243-3252.
- University of Michigan. (2018). Inverse Kinematics. Available at: http://web.eecs.umich.edu/~ocj/courses/autorob/autorob_10_ik_closedform.pdf [Accessed 01 Dec. 2018].
- Chris Kirtley. (unknown). Inverse Dynamics. Available at: http://www.clinicalgaitanalysis.com/teach-in/inverse-dynamics.html. [Accessed 01 Dec. 2018].
- Michael Sherman. (2011). Simbody documentations. Available at: https://simtk.org/docman/?group_id=47. [Accessed 01 Dec. 2018].
- NCSRR. (2018). Opensim documentations. Available at: https://simtk-confluence.stanford.edu/display/OpenSim/Documentation. [Accessed 07 Dec. 2018].